Archivo de la categoría: Curiosidades microbiológicas
Escherichia coli , ¿nuestro futuro surtidor de gasolina?
Así es queridos lectores, el artículo publicado nada más y nada menos que en Nature y titulado ‘Producción microbiana de los alcanos de cadena corta’ , llevado a cabo por científicos surcoreanos dirigidos por el profesor Sang Yup Lee, afirma que han sido capaces de producir combustible a partir de Escherichia coli intestinales modificadas genéticamente. Prometedor, ¿verdad?
Mediante modificaciones genéticas, se manipuló el metabolismo de ácidos grasos de Escherichia coli para que proporcionaran derivados de ácidos grasos más cortos que los que usualmente producen, introduciendo así una nueva ruta totalmente sintética para la biosintesis de alcanos, que son hidrocarburos de cadena corta.
¿Cual es la importancia de este descubrimiento? La gasolina, el producto derivado del petróleo que es el más ampliamente utilizado como un combustible para el transporte, es una mezcla de hidrocarburos, aditivos y agentes de mezcla. Los hidrocarburos, llamados alcanos, consisten sólo en átomos de carbono e hidrógeno. La gasolina tiene una combinación de alcanos de cadena lineal y cadena ramificada que se componen de 4-12 átomos de carbono unidos por enlaces directos carbono-carbono.
Anteriormente, a través de ingeniería metabólica de E. coli, se produjeron algunos resultados de la investigación sobre la producción de alcanos de cadena larga, que constan de 13-17 átomos de carbono, adecuados para la sustitución de diésel. Sin embargo, no se ha informado hasta ahora de la producción microbiana de alcanos de cadena corta, un posible sustituto de la gasolina.
Editado (29/10/2013): «Producir hidrocarburos con bacterias no es un tema trivial. No solo se requiere de las rutas de síntesis de alcanos en una bacteria, sino que además, y aquí está lo complicado realmente porque alcanos producen muchas bacterias, hay que redirigir el flujo de Carbono hacia la síntesis de este tipo de compuesto casi en exclusiva, sin comprometer del todo la supervivencia de la bacteria. Es decir, hay que hacer que el sistema se pueda activar en un determinado momento, cuando tengamos una masa crítica de bacterias. Además estas bacterias deben soportar la presencia del compuesto. NO solo deben producir gasolina, sino que además deben vivir a gusto en gasolina» @scariosHR
¿Conseguiremos en el futuro dejar de abusar del petróleo y tener granjas de bacterias produciendo gasolina? ¿No habrá ni que repostar porque tendremos colonias de bacterias produciendo gasolina directamente en el depósito de nuestro vehículo?
Pues no me extrañaría, pero todo parece indicar que ese futuro, por desgracia para nuestro planeta, es más bien lejano, ya que el equipo encargado del experimento ha logrado obtener, de momento, 580 miligramos de gasolina por cada litro de aceite bacteriano producido. Tienen como objetivo conseguir una tasa competitiva de unos 20 gramos por litro de aceite. ¿Lo conseguirán? Eso espero.
A modo personal, considero que estamos ante una auténtica revolución bacteriana, cada día son más los experimentos que usan como fábricas a las bacterias, para producir todo tipo de compuestos, antibióticos, anticuerpos, gasolina, alimento…sin duda el potencial de estos organismos de ser modificados genéticamente para producir compuestos elegidos por el experimentador, es enorme.
Sin duda como ya he comentado anteriormente, lo que mueve a una especie es su supervivencia, y la nuestra actualmente está amenazada por nuestro modo de vida y nuestra manera de consumir y obtener recursos. Por está razón he elegido comentaros hoy está utilidad de los microorganismos frente a muchas otras que me llaman la atención, porque considero que para poder seguir descubriendo, debemos poder seguir habitando este planeta, y para que eso suceda, debemos cambiar nuestro estilo de vida, y el uso de combustibles actuales está sin duda dañando seriamente el equilibrio dinámico que permite la vida.
Debemos comenzar con entender que el planeta y sus recursos no nos pertenecen, que pertenecemos nosotros a ellos. El ser humano consume cerca de un 40% de todos los recursos terrestres que produce el planeta. Se conocen alrededor de 1,75 millones de especies, si solo una especie consume el 40% de los recursos terrestres…¿que estamos dejando para los demás organismos? Organismos que para más abuso, utilizamos en cuanto sabemos como hacerlo.
A menudo pienso las prioridades que deberíamos establecer, por ejemplo, yo soy un apasionado de la biomedicina, pero…¿para que queremos la medicina si en 50 años, por decir una cifra al azar, estamos condenados a la extinción por nuestro estilo de vida? ¿Debemos establecer claras prioridades en las investigaciones? ¿Se debe comenzar por cambiar nuestro estilo de vida, asegurando que nuestra existencia no es nuestra condena, para después tratar de potenciar otras áreas de la ciencia?
Estas preguntas me vienen a diario a la cabeza, como estudiante con vocación investigadora debo decidir donde acabo investigando, si es que algún día consigo investigar, y el hecho de saber que puedo enmarcar mi carrera en distintas áreas, y no saber cual es la más necesaria, es un gran dilema, porque por norma general, una vez elegida un área, en esa área te quedas, cosa que comprendo, pero que a la vez, proporciona inseguridad a la hora de decidir.
Espero que les haya gustado el texto, y perdonen como ha ido derivando, pero creo que el blog está hecho para eso, no simplemente informar, si no conseguir transmitir las reflexiones producidas por la obtención de información.
Javier Fernández.
Schizosacchromyces pombe, la levadura que rejuvenece cuando se reproduce.
Como especie, algo que siempre me ha llamado la atención es que nos consideremos superiores a cualquier otra especie. Es decir, tenemos la capacidad, la potente capacidad de observar nuestro mundo y poder comprenderlo y entenderlo, tenemos eso que solemos llamar inteligencia… ¿y acaso la demostramos? En el momento en que, entendiendo las complicaciones de la vida, de las formas vivas, nos consideramos superiores y más importantes que las demás especies, en mi opinión, perdemos cualquier ápice de inteligencia que creyésemos que nos quedaba.
Algo que la sociedad humana a deseado desde su existencia, es la capacidad de evitar el envejecimiento, e imaginaros que lo conseguimos tal y como lo ha hecho Schizosacchromyces pombe, y que cada vez que nos reprodujéramos, rejuveneciéramos. Eso tendría sus ventajas y desventajas, las ventajas las veo tan directas que las vamos a obviar, pero la desventaja de la sobrepoblación es muy remarcable. Vaya, ojalá tuviéramos el tamaño de S. pombe y ese no fuera un gran problema.
Esta curiosa levadura, ha conseguido volverse inmune al paso del tiempo. Solo necesita que las condiciones de crecimiento sean especialmente favorables, y conseguir reproducirse. Por norma general, las levaduras y muchos otros microbios, se dividen mediante escisión o bipartición, y aunque al principio se creía que los dos descendientes resultantes de la división eran exactamente iguales, más tarde se demostró que uno de los dos microbios descendientes del original, recibe material más viejo, y que el otro recibe un material totalmente nuevo, completamente funcional, como si renovase su maquinaria por completo, o como si a nuestro coche, cada 10.000 kilómetros, le cambiáramos el motor y la carrocería. Pero el problema es que si un organismo, que desaparece al reproducirse, da lugar a solo un organismo “funcional”, la población no podrá crecer nunca.
En condiciones especialmente favorables, se ha demostrado que S. pombe, al reproducirse, entrega material viejo de igual manera a los dos descendientes, haciendo que tras cada división, los descendientes sean un poco más jóvenes que su predecesor, porque la mitad de su material es nuevo, a diferencia del material del organismo anterior, que tras el uso se había ido desgastando. Es decir, de cada división no sale un solo organismo con material nuevo, y otro con material viejo, si no que salen dos con material parcialmente nuevo, lo que permite una mayor adaptación y posibilidad de supervivencia a ambos organismos, y no solo al que se le había otorgado el material nuevo.
A diferencia de otras especies, S. pombe puede escapar al envejecimiento, siempre y cuando se divida lo suficientemente rápido, por eso debe estar en condiciones especialmente favorables, y no solo en condiciones normalmente favorables. ¿Pero qué ocurre cuando vive en condiciones que no sean perfectas? Para averiguarlo, los investigadores expusieron la levadura al calor, la radiación ultravioleta y productos químicos perjudiciales, lo que retrasó su crecimiento a un punto en el que los microbios no podían dividirse lo suficientemente rápido para mantenerse jóvenes. Bajo estas condiciones negativas, el asombroso elixir de la juventud del que parecía disponer S. pombe, parece que se termine, ya que las células de levadura comenzaron a dividirse en una más joven y otra más vieja, al igual que lo hacen por norma general el resto de microbios.
«Es increíble ver que incluso esos organismos simples han desarrollado estrategias tan potentes para sobrevivir», dijo Thilo Gross, y de nuevo, podemos volver a la pregunta que establece la pequeña reflexión del primer párrafo. ¿Somos superiores al resto de especies, tal y como por norma general se cree? Nosotros, los Homo sapiens sapiens (encima dos veces, a modo mofa, no somos sabios, somos sabios sabios), con toda nuestra supuesta inteligencia, no hemos sido capaces de crear estos mecanismos por nosotros mismos, y una “simple” levadura lo ha conseguido. ¿Debemos dejar de creernos mejores, y valorar a cada especie por igual? Al fin y al cabo, todas han llegado a donde estamos nosotros, a estar vivos en esta época, pero, a diferencia de nosotros, muchas de esas que consideramos “simples” especies, llevan aquí mucho más tiempo que nosotros, y seguirán aquí mucho más tiempo después de que nosotros desaparezcamos, si el objetivo final de una especie es su supervivencia, y tratar de evitar la extinción… ¿Qué especie es mejor? ¿Una levadura o un humano? ¿Un artrópodo o un chimpancé? Entiendo que nos podamos creer superiores porque no se puede negar que somos más complejos que las demás especies, pero parece que cuando se trata de sobrevivir, ser complejo no es una ventaja.
No pretendo ser, para nada, taxativo en mis afirmaciones, y aunque defiendo mi postura anterior, también agradezco a la complejidad permitirme anonadarme con cada cosa que descubro, y que la complejidad de mi ser me permite descubrir. Quizá como especie no debamos agradecer esa complejidad, pues sin duda, nos llevará a la extinción, pero como organismos, estoy seguro de que podemos estar orgullosos de ser complejos.
¿Será cuestión de límites?
Entendiendo un Western Blot (o al menos, intentándolo)
Pues después de pasar el tramo de los exámenes finales, y el inicio del verano, y con motivo del gran Carnaval de Química que está organizando @Luisccqq, y del que quiero formar parte, volvemos a la carga. Desde que acabé las clases estoy colaborando por puro placer en el laboratorio de Biomedicina molecular y celular de la Universidad de las Islas Baleares (UIB). Si algo he aprendido en estos 7 días que llevo paseándome por el laboratorio y ayudando en lo que puedo, es que hay algo que es de vital importancia controlar, hacer un Western Blot, y como autoayuda a mi mismo y a vosotros, vamos a tratar de explicar que es, en que se basa, y como se prepara, de la forma más clara posible.
Esto NO ES UN PROTOCOLO, simplemente vamos a indicar los pasos que se siguen, que se usa, y en que se basa, centrándonos en esto último, poder explicar el fundamento teórico de todo lo que, siendo estudiante de tercero de biología, pueda comprender. Al final de la entrada os dejaré una recopilación de protocolos, pero prefiero dedicar esta entrada a que entendáis que es un Western Blot, y si queréis hacer uno, uséis el protocolo de vuestro laboratorio. En algunos pasos ahí un protocolo, pero no es más que para que veáis lo que se usa y porque, y he ido mezclando diferentes protocolos, así que NO SIGÁIS LOS PROTOCOLOS.
¿Que es un Western Blot?
Desarrollada en el laboratorio de George Stark, en Stanford, es una técnica analítica que se usa para detectar y cuantificar proteínas especificas en una muestra determinada, una muestra que por norma general tiene una gran cantidad de proteínas, unas que nos interesarán, y otras que no.
Básicamente, lo que más adelante profundizaremos, se puede resumir en realizar una electroforesis que nos separa las proteínas según un determinado criterio, una transferencia del gel resultante, a una membrana adsorbente (proceso por el cual átomos, iones o moléculas son atrapadas o retenidas en la superficie de un material). y posteriormente, se detecta la proteína de interés , y se estudia la unión antígeno-anticuerpo, y se observan los resultados, viendo si hay más, si hay menos, si ha cambiado su configuración…
¿Que necesitamos saber, para entenderlo?
Pues que leáis poquito a poquito los párrafos inferiores, con calma y paciencia, y si algo no os queda claro, aunque lo haya intentado, tirad de Interné, ese gran sabelotodo. Y un pequeño glosario:
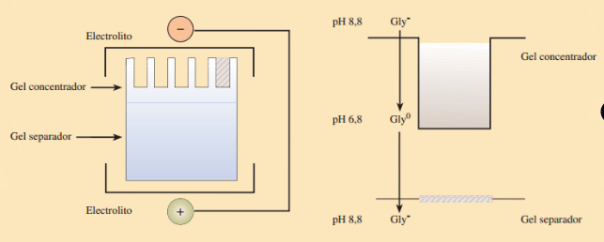
Electroforesis discontinua: Electroforesis donde hay dos tipos de geles, un gel concentrador (stacking) de poros grandes, y un gel separador (running), de poros pequeños.
PVDF: Fluoropolimero inerte químicamente, se usa en Western Blot porque es afín a las proteínas, más concretamente a los aminoácidos.
Anticuerpo: Inmunoglobulinas. Son glucoproteínas que neutralizan elementos externos (antígenos), como bacterias, parásitos, virus…cada anticuerpo es único y específico para cada tipo de antígeno.
Antígeno: Proteínas o polisacáridos. Sustancias externas que desencandenan la formación de anticuerpos y puede causar una respuesta inmunitaria mediante la activación de linfocitos.
PBS (Tampón fosfato salino): Solución acuosa y salina que contiene cloruro de sodio, fosfato de sodio, cloruro de potasio y fosfato de potasio. Se usa principalmente para mantener un pH estable-
SDS: Dodecil sulfato de sodio. Detergente de carga negativa. Se une a las proteínas y les confiere dicha carga. Esta unión se da en una proporción constante, cada gramo de proteína se une a 1,4 g de SDS, sea cual sea la proteína.
Anticuerpo primario: Se une y marca la proteína de interés. 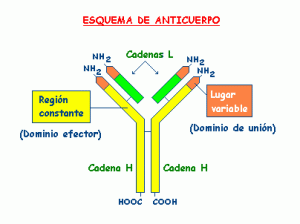
Anticuerpo secundario: Determinan si el anticuerpo primario se unió a la proteína de interés, los hay de diversos tipos según como los queramos o podamos detectar luego: Ligados a enzimas (veremos un cambio de color), de marcado radiactivo, de marcada fluorescente (emitirá fluorescencia..)
Anticuerpo monoclonal: Los anticuerpos monoclonales son anticuerpos idénticos, porque son producidos por un solo tipo de célula del sistema inmune. Se unen a un epítopo en partícular, una única región del antígeno.
Anticuerpo policlonal: Son anticuerpos derivados de diferentes tipos de linfocitos. Los anticuerpos policlonales son una mezcla de inmunoglobulinas secretados en contra de un antígeno concreto.
¿Cómo se prepara y realiza un Western Blot, y en que se basa?
Paso 1. Preparación de la muestra.
Lo primero de todo, como es obvio, es disponer de una muestra, ya sean muestras tomadas de un tejido (cerebro de rata, por ejemplo), o de un cultivo celular directamente. Debemos asegurarnos que en dicha muestra tenemos una cantidad de proteínas suficientemente alta como para poder trabajar.
¡Trabajamos siempre en eppendorfs!
Debemos coger la muestra, y lo primero que se hace es introducirla en un Buffer de Extracción. Cuando buscamos purificar y estudiar proteinas, lo primero que se debe hacer es romper las células , y extraer de el la fracción de proteina. Todo esto con el buffer de extracción. Es un proceso importante, ya que los métodos que se usan, tanto físicos como químicos, si no se hacen bien, pueden afectar a la integridad de la proteína, degradarla, y en cualquier caso, afectar a nuestros resultados.
También pueden usarse detergentes, sales o tampones que favorezcan la lisis y solubilicen las proteínas. Es muy recomendable añadir al buffer de extracción inhibidores de proteasas y fosfatasas que evitan la digestión por las enzimas celulares de las proteínas y de las fosfoproteínas, respectivamente, y si no los usamos, corremos el riesgo de perder la muestra deseada.
Lo primero es lavar la muestra con PBS, y se resuspende en un volumen «x», que para tener concordancia con lo de abajo, diremos que es 250 uL de:
- Buffer de extracción, que lleva (para 25 mL)
- Tris-HCl 50mM: Como tampón.
- NaCl 120 mM
- IGEPAL al 0,5%. : Detergente que ayuda a la lisis de las células.
- Ph 8.0
- NaF 100 mM : Antifosfatasa, la fosfatasa es una enzima hidrolasa responsable de eliminar grupos de fosfatos de varios tipos de moléculas como nucleótidos, proteínas…y no nos interesan que nos toques las proteínas, que las hemos extraído nosotros pa’ algo.
- NaVO3 200 uM : Proporciona actividad higroscópica, que es la capacidad de algunas sustancias de absorber humedad del medio circundante.
- Antiproteasas (1 pastilla en 2 ml de agua, que se añaden a estos 25 mL totales) , y que creo que no hace falta decir para que se usan…(shhhh, que se cargan las proteínas…)
Después se homogeniza la muestra, ya sea con un homogenizador, o sonicando, si es así…¡recordad usar cascos! Después de la homogeinización, debemos centrifugar, por norma general a 13.000 rpm, durante 10 minutos, y a 4ºC, que ayuda a conservar mejor el material biológico.
Nota: Recordad que la sonicación es el acto de aplicación de la energía del sonido, generalmente ultrasonido, para agitar las partículas de una muestra, por ello he dicho que es recomendable usar cascos, por el bien de vuestros tímpanos.

Sonicando, por http://jindetres.blogspot.com.es/
Después de tener nuestro pellet (En los procesos de centrifugado, se denomina «pellet» al material sedimentado), debemos, con sumo cuidado para no afectar al pellet, vaciar el eppendorf, sin brusquedades, en un papel limpio y dejarlo abierto para que seque bien, no debe quedar nada del buffer de extracción.
Paso 2. Cuantificación de proteínas
¿Porqué cuantificar proteínas? Es muy simple, cuando más adelante debamos cargar proteínas, para que los resultados tengan algo de sentido, tendremos que saber cuanta proteína tenemos, para cargar lo mismo y que los resultados no dependan de cuanta proteína hemos cargado, si no de las características de la proteína cargada.
¿Cómo se hace? Por norma general se usa «BCA® Protein Assay Kit» , un kit que ya viene preparado (y que como todos los kit´s, no sabes que llevan para que no lo puedas preparar y te veas obligado a comprarlo a la casa).
Como este paso con el kit es siempre igual (que yo sepa), en este caso si os voy a poner un protocolo, que está aquí.
Hacemos todas esas diluciones entre en la curva estándar que tiene que tener vuestro laboratorio, donde veréis que longitud de onda corresponde a que cantidad de BCA, y extrapolando con vuestra longitud de onda, y teniendo en cuenta las diluciones que habéis hecho, sabremos que cantidad de proteína tenemos.
Así ya sabemos la cantidad de proteína que tenemos en nuestras muestras, y jugando con disoluciones, podremos cargar la cantidad deseada de proteína siempre que queramos.
Paso 3. Electroforesis en gel.
La función de la electroforesis es separar, basándose en uno o varios criterios, las proteínas. Estos criterios suelen ser: peso molecular, punto isoeléctrico, y carga eléctrica. Si solo se usa un criterio, estamos ante un gel en 1 dimensión, si por ejemplo, se combinan en el mismo gel, el peso molecular y el punto isoeléctrico, estamos ante un gel bidimensional.
La electroforesis más común, que no la única, es la SDS-PAGE (polyacrilamide gel electrophoresis), que usa gel de poliacrilamida, y SDS, que es un tampón de dodecilsulfato, que no es más que un detergente, que actúa rompiendo enlaces no covalentes en las proteínas, desnaturalizandolas, y obligando a las proteinas a adquirir su conformación primaria, lo que permite una mejor clasificación sin interferencias debidas a las posibles conformaciones (que recordemos que hay hasta cuatro).
El SDS lo que hace es unirse a las zonas apolares de un polipéptido, lo que proporciona al polipéptido una carga negativa que resulta proporcional al número de aminoácidos, y es lo que acaba por obligar a la proteína a adoptar su conformación primaria.
Las proteínas presentan una carga eléctrica neta si se encuentran en un medio que tenga un pH diferente al de su punto isoeléctrico y por eso tienen la propiedad de desplazarse cuando se someten a un campo eléctrico. La velocidad de migración es proporcional a la relación entre las cargas de la proteína y su masa. Cuanto mayor carga por unidad de masa más rápida será la migración. Una ventaja importante de los geles de poliacrilamida es que son químicamente inertes, transparentes y estables en un rango amplio de pHs, temperatura y fuerza iónica.
Cosas que debemos tener en cuenta cuando hacemos una electroforesis con geles de poliacrilamida:
a) Los geles de poliacrilamida se forman por la polimerización de la acrilamida por acción de un agente
entrecruzador (‘cross-linking’), la bis-acrilamida en presencia de un iniciador y un catalizador. Como
iniciador se suele utilizar TEMED (N,N,N,N’-tetrametilnediamina) y como catalizador el ión persulfato
(S2O8-) que se añade en forma de persulfato amónico (APS). En algunas situaciones, como por ejemplo en el isoelectroenfoque en el que la presencia de persulfato puede interferir con la electroforesis se emplean
riboflavina y TEMED.
¿Que componentes lleva y porqué?
- Tris como tampón, es inocuo para las proteínas.
- Glicina como fuente de iones de arrastre, o lentos, se sitúa en un valor inferior a la de la proteína mas lenta conocida, con carga negativa.
- Acrilamida, que en disolución acuosa autopolimeriza de forma espontánea y lenta, y en presencia de un sistema generador de radicales libres, los monómeros de acrilamida se activan, y reaccionaran para formar polímeros de larga cadena. Estos polimeros no forman un gel, son viscosos, debido a que las cadenas pueden deslizarse unas sobre otras, por eso se utiliza….
- Biscarilamida, polimeriza conjuntamente con la acrilamida pero establece puentes entre las cadenas lineales de acrilamida, evitando el deslizamiento de las anteriores, y produciendo de esta manera la formación de gel.
- SDS, explicado arriba.
- APS, Persulfato amónico, que genera radicales libres, y es lo que como ya hemos dicho antes, inicia la reacción de polimerización de acrilamida, que lleva finalmente a la formación del gel.
- TEMED, N,N,N’,N’-tetrametiletilenodiamina, es otro iniciador. La tasa de polimerización y las propiedades del gel resultante dependen de la concentración de APS y TEMED. Aumentando su contenido se disminuye la longitud media de la cadena de polímero y se incrementa la turbidez del gel, al tiempo que disminuye su elasticidad. Por contra, disminuyendo la cantidad de iniciadores se obtiene el efecto inverso. Se debe, por tanto, utilizar la menor concentración posible de catalizadores que permita la polimerización en un tiempo óptimo.
b) Las soluciones de acrilamida se desgasifican pues el oxígeno es un inhibidor de la polimerización. Además, durante la polimerización se libera calor que podría provocar la formación de burbujas en el seno del gel.
¿Cómo desgasificamos? Muy simple, añadimos toda el agua que podamos (evitando que rebose), que además mantendrá hidratado al gel, pero recordad, ¡usad agua bidestilada!
c) La velocidad de polimerización viene determinada por la concentración de persulfato (APS – catalizador) y TEMED (iniciador).
d) La porosidad del gel la determina las proporciones relativas de poliacrilamida y bis-acrilamida, siendo menor el poro cuanta más bisacrilamida vs. acrilamida se use.
e) El porcentaje total de acrilamida/bisacrilamida determina el rango de separación del gel. Habitualmente los geles se denominan en función del % de acrilamida/bisacrilamida que contienen. Así, la mayoría de las proteínas se separan bien en el rango de 5 a 10%. Un menor porcentaje (mayor tamaño de poro) es mejor para separar proteínas de gran tamaño.
Por norma general se emplean sistemas de dos tampones, conocido como sistema discontinuo. Este sistema permite la separación de volúmenes relativamente grandes de muestra sin pérdida de resolución. Usaremos dos geles, que llevan los mismos componentes, pero en diferente concentración, y cada uno de ellos puede estar en un porcentaje determinado, dependiente de la cantidad de proteína que disponemos, yo como novato no se que relación se sigue, así que si tenéis jefe, preguntadselo a él, y si no…enhorabuena, sois jefes. Por poner un ejemplo, y digo un ejemplo porque cada laboratorio es un mundo:
En los sistemas discontínuos el primer gel, que es el Running, asegura la migración de todas las proteínas en el frente de migración, provocándose la acumulación de todas las que se han cargado en el pocillo. La separación realmente comienza a partir del momento en el que el frente de migración alcanza la frontera del segundo gel, el stacking. El gel que queda al final en la parte superior (‘stacking’), es de mayor poro (menor porcentaje de acrilamida+bisacrilamida) y tiene un pH más ácido que el segundo gel que es el que realmente separa las proteínas. Como ya hemos dicho este sistema es especialmente adecuado para analizar muestras diluidas sin perder resolución.
Para la realización de la electroforesis, tras preparar los geles de poliacrilamida, al porcentaje que necesitéis, que será el medio donde colocaremos la proteína de nuestra muestra, debemos colocarlos en la unidad de electroforesis, que si es la primera vez que la preparais, es así:
Para solo un gel:
Más moderno, con 4G y para dos geles:
Se prepara el tampón de electroforesis (tris/glicina) y se añade en los reservorios interior y exterior, y debemos retirar las posibles burbujas de aire del interior de los pocillos (vamos, como ya hemos dicho antes, ¡a llenarlo de agua!)
Con la ayuda de una micropipeta cargamos las proteínas de nuestra muestra (¡antes del paso I!), y recordadlo, seguid un orden que conozcáis.
¿Dónde se cargan las proteínas? Una vez colocados los dos geles, antes de echar el buffer/tampón tenemos que colocar en el gel concentrador, un peine (ver paso G), que nos creará unos pocillos en dicho gel, que será donde cargaremos nuestra proteína.
En el primer pocillo (aunque podéis hacerlo donde queráis), añadid un marcador de peso molecular, para después saber el peso molecular de vuestras muestras, observando el marcador y vuestra muestra, y la distancia que han recorrido tras la electroforesis.
Se conectan los electrodos a la cubeta de electroforesis, y a la fuente de alimentación, y conectamos la corriente eléctrica, recordad antes de todo que los electrodos están correctamente colocados, cada color con su color, rojo con rojo y negro con negro, y recordad que el voltaje es variable dependiendo del espesor del gel, de la cantidad de proteína…pero que nunca debe bajar de 100V, y se deja correr hasta que el frente azul, que son nuestras proteínas, llegan a la parte inferior del gel, y nada más sacarlo, recordad recortar una esquina del gel para saber en que posición estaba orientado, y así sabremos que muestra hay en cada pocillo, porque…¡antes lo habéis apuntado! o…más os vale tener buena memoria.
Finalizada la electroforesis, sacados los geles, y marcados para saber su orientación, pasamos al punto 3.
Paso 4. Tinción del gel
Simplemente se coloca el gel, en un «tupper» (para entendernos), con un colorante de proteínas, el Azul de Coomassie (bueno, con sensibilidad hasta 50 ng) u otro igualmente válido, que nos dirá, según la intensidad, la cantidad de proteína existente (de forma cualitativa), o si directamente no existe proteína. El Azul de Coomassie es de tipo aniónico y se une a proteínas de forma inespecífica.
Debemos dejar el gel, junto a la solución de Azul de Coomassie (anexo), de un volumen de diez veces el del gel, en agitación suave durante unos 30 minutos aproximadamente, tras los cuales, podremos recoger con la ayuda de una pipeta la solución, y almacenarla para futuras tinciones.
Con el objetivo de limpiar mejor el gel, y que no quede teñido de azul por todos sitios, si no solo el los lugares en los que hay proteínas, se deja de nuevo en agitación suave el gel junto a una solución para desteñir, que se debe cambiar tantas veces como sea necesario, es decir, cada vez que veamos que la solución para desteñir a pasado de ser incolora a azul (lo que indica que ha limpiado el gel), y este proceso puede durar el tiempo deseado, hasta obtener el resultado espero. En mi caso, esta solución para desteñir, la preparo con:
- 925 mL de agua destilada MiliQ, o bidestilada.
- 50 mL de Metanol.
- 75 mL de Ácido Acético puro.
¡Recordad hacerlo en la campana, que el acético te deja KO! Y también, recordad que debéis echar el metanol y el acético en los 925 mL de agua, y no al revés, por motivos armamentísticos…ya sabéis…¡BUM!
Pasado el tiempo necesario para la destinción, limpiamos el gel con agua destilada, y si es necesario, ya podemos conservar el gel a 4ºC durante un largo periodo de tiempo, si fuese necesario esperar antes de proseguir con el siguiente paso, la transferencia.
Quedará algo así:

Paso 5. Transferencia.
Paso que permite que las proteínas sean accesibles a la detección por anticuerpos. Simplemente, los resultados son mucho mejores, y desde siempre se ha hecho así, se se transfiere a las proteínas desde el gel de poliacrilamida, a una membrana que, normalmente, es de nitrocelulosa o PVDF. En las membranas de nitrocelulosa, las proteínas interaccionan con esta mediante enlaces no covalentes de naturaleza hidrófoba. Por otro lado, en las membranas de PVDF, resulta que el fluoruro de polivinilideno, inerte químicamente, es afín a las proteínas, mediante enlaces hidrofóbicos y de dipolos membrana – proteína.
Las dos opciones son igual de válidas, pero debéis saber que las de PVDF son más caras, por el hecho de ser más resistentes, no son tan fáciles de quebrar como las de nitrocelulosa, y pueden someterse a varias pruebas consecutivas.
La transferencia puede hacerse mediante tres métodos: Difusión, vacío, o electrotransferencia, este último es el más usado con diferencia, y el que mejores resultados ofrece, pero no viene mal explicar los otros métodos y así sacamos algo de «Historia del Western Blot».
Transferencia por difusión: Su propio nombre lo indica, simplemente se ponen en contacto el gel y la membrana, y por simple difusión, se transfiere. Se ponen en contacto entre sí y con un tampón, que asciende por capilaridad hacia el papel de filtro, que se habrá colocado encima de la membrana, y mientras lo hace, arrastrara a las proteínas hasta la membrana, donde quedarán retenidas.
La cantidad de proteína transferida no es muy alta, aunque es funcional, pero está siendo remplazado por los otros métodos, aunque una modificación está dándole de nuevo algo de uso, ya que permite a partir de un mismo gel, transferir el contenido a varias membranas, pero esto solo sirve si no hace falta cuantificar proteínas, y ya os digo yo, que esto no suele ser lo común.
Transferencia al vacio: Al método anterior, se le añade el poder de succión de una bomba, que se conecta a un sistema de secado de planchas de gel, que lleva a as proteínas desde el gel hasta su retención en la membrana. Interesante porque la cantidad de proteina transferida es mayor, y válido tanto para proteinas de alto, como de bajo peso molecular.
Electrotransferencia: La que vais a usar, con diferencia la más usada y la más recomendada. Al igual que durante la realización de la electroforesis, se usa una corriente eléctrica y un tampón (Buffer de transferencia), que rellena la cubeta de electrotransferencia, como resultado se transfieren las proteínas. En este caso, se usan los siguientes elementos, que se colocan partiendo desde el polo negativo (cátodo, ¡rojo!) al positivo (ánodo, ¡azul!):
- Esponja
- 2-3 papeles de filtro Whatman nº3 empapados en buffer de transferencia
- Gel
- Membrana
- 2-3 papeles de filtro Whatman nº3 empapados en buffer de transferencia
- Esponja
A este conjunto se le conoce como Sandwich, y una vez montado, debéis meterlo en la cubeta con el negro del sandwich (no la parte torrada del sandwich que traéis de casa), orientado hacia el polo negro de la cubeta.
Al conectar las corriente, a una determinada potencia que depende de diversos factores (que no debe bajar de 100V), las proteínas se desplazarán hacia el polo positivo, y quedarán retenidas en la membrana.
Os informo de que hay dos tipos de electrotransferencia:
Húmeda: La que yo he usado todas las veces que he hecho electrotransferencia, así que ante las dudas ya sabéis. Para que podáis ver la diferencia que hay entre los dos métodos, aunque no queria y lo estoy haciendo, os pongo un ejemplo de como varia el buffer de transferencia en ambos casos:
- Tris base 25 mM
- Glicina 192 mM
- pH 8,2
- 20% de metanol (v/v)
Semi-seca: Es más rápida que la anterior, pero no se recomienda para proteínas mayores a 100kDa. Cómo podréis ver, es más ligera (menos glicina) y permite que las proteínas corran más rápido y se transfieran antes, pero no siempre funciona, así que limitaros a usarla cuando os lo digan o cuando esteis seguros de que vuestras proteínas pesan menos de 100 kDa.
- Tris base 50 mM
- Glicina 40 mM
- 0,04% de SDS (p/v)
- 20% de metanol.
Quedará una copia del gel, pero en la membrana, algo así (como véis, ya no es gel):
Paso 6. Bloqueo.
Con este paso simplemente se trata de bloquear la unión de proteínas a la membrana, que puedan unirse a esta en los lugares que hayan quedado libres, posteriormente a la transferencia. Sin este paso, el anticuerpo, que es de naturaleza proteica, podría unirse en esos huecos libres (recordemos que la membrana es afín a las proteínas), y dificultar la distinción del complejo antígeno-anticuerpo que se trata de obtener.
Para bloquear, se suele usar una solución proteíca, que incluye albúmina de suero bovino (BSA), leche en polvo o caseína, y algo de detergente, como Tween 20. Esta solución proteíca rellena los huecos libres, y como el anticuerpo que se usará es específico, no se unirá en estos huecos,ya que no habrá en ellos la proteina que el anticuerpo busca, con lo cual no nos molestarán para los siguientes pasos.
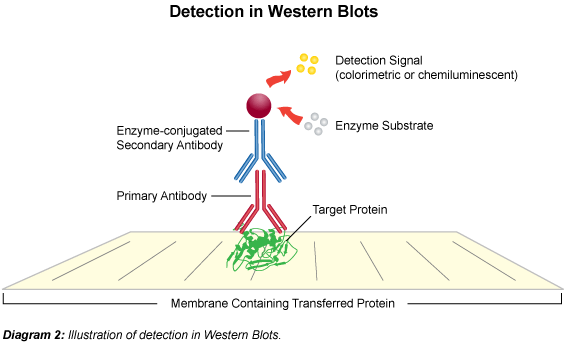
Paso 7. Detección.
Se trata de buscar en la membrana una determinada proteína, y por este mismo motivo se usa un anticuerpo específico, unido a una enzima, que si encuentra su sustrato ( la proteína indicada, o más bien su antígeno), se producirá una reacción y podremos observar a la proteína por diversos métodos. Hay muchos procesos que llevan a una detección, voy a mencionar el más famoso o conocido, o que directamente…utilizo yo y considero más molón.
Detección en dos pasos: ¡El de toda la vida! Se tiene que usar un anticuerpo primario y un anticuerpo secundario.
Anticuerpo primario
El que se une a la proteína buscada. El anticuerpo se consigue inoculando la proteína a un animal, que puede ser casi cualquiera de los que vemos en un zoo, conejos, monos, o, mucho más ecológico, procedente de un cultivo celular. Esto desencadena en el animal una respuesta inmune, produciendo así un anticuerpo, primario, porque reconoce la proteína.
Cogemos la membrana que habíamos bloqueado y se deja en agitación moderada junto a una disolución, pequeñisisisisisisima, de anticuerpo primario, que solo tiene desde 0,5 a 5 ug/ml, y se usan microlitros. Los anticuerpos se guardan en un congelador a -20ºC, por norma general, y son caros, así que más vale que no os vean malgastando alguno.
Cogemos el mismo tupper que teníamos, previo lavado, y juntamos la membrana junto a la ya mencionado disolución, que lleva cloruro de sodio, algo de detergente (Tween 20), y en ocasiones BSA o otras proteínas. Se deja «incubando» desde media hora hasta «Overnight«, es decir, durante toda la noche.
Lavado del anticuerpo primario.
Para lavar la membrana y dejarla limpia de anticuerpo primario, se puede usar TBS, 2 lavados de 30 segundos. El TBS ( o Buffer salino de Tris), se prepara de forma muy simple con:
- 50mM Tris.
- 150 mM NaCl
- Ajustar pH con HCl hasta 7,6.
La forma más simple de prepararlo es usar unas tabletas ya preparadas de TBS, que vienen formuladas para diluirlas en 500 ml de agua desionizada.
Puede usarse también T-TBS, que no esmás que TBS al que se le añade Tween 20, un detergente, que ofrece mejores resultado. Se prepara:
- 50 mM Tris, un tampón ácido – base.
- 150 mM NaCl
- 0,05% Tween 20, un detergente que ayuda a realizar una limpieza mejor.
- Ajustar pH con HCl (diluido, si no el pH bajará de forma drástica) hasta 7,6.
Cabe indicar que también venden pastillas ya preparadas con esta formulación para ser diluidas en 500 mL de agua desionizada.
Anticuerpo secundario.
Tras lavar la membrana para eliminar el anticuerpo primario que no se ha unido, se expone al anticuerpo secundario. Éste reconoce de forma específica una región concreta del anticuerpo primario, a la cual se une, y estos secundarios suelen estar marcados para ser detectables por diversos métodos (unión a biotina, a una enzima «reporter» como la fosfatasa alcalina, etc.).
Varios de estos anticuerpos se unirán a cada anticuerpo primario, por lo que acaban resultando en una amplificación del anticuerpo primario, y no solo eso, si no que ahora podemos detectar al anticuerpo primario. Un anticuerpo secundario «anti-ratón» es aquel capaz de reconocer casi todos los anticuerpos primarios obtenidos de ratones. Y así con el resto de anticuerpos secundarios «anti-(inserte animal)», «anti-cabra», «anti-conejo»… cabe indicar que pueden emplearse también proteínas y no anticuerpos, como las proteínas A y G.
Lavado del anticuerpo secundario.
Para lavar la membrana y dejarla limpia de anticuerpo secundario, pueden darse 3 lavados de 3 minutos de T-TBS, que no es más que TBS al que se le ha añadido Tween 20. (explicada su preparación arriba)
Paso 8. Análisis
Simplemente, dependiendo del tipo de anticuerpo secundario que hayamos usado, lo analizaremos de una determinada forma. Os recomiendo ver el glosario de nuevo:
Análisis colorimétrico
Considero que el más sencillo. La detección colorimétrica depende de la incubación del Western blot con un sustrato unido al anticuerpo secundario que es una enzima «reporter» , (una peroxidasa, por ejemplo) , produciéndose una reacción que acaba en un cambio de color. La cuantificación proteica se evalúa por densitometría (intensidad o densidad de la proteína, de lo que vemos de proteína, de la «mancha») o por espectrometría, que nos indicará un determinado valor de longitud de onda, equivalente a una determinada cantidad.
Análisis quimioluminiscente
La detección quimioluminiscente requieren la incubación de la membrana con un sustrato que trae unido el anticuerpo secundario, que emitirá luminiscencia al ser expuesto al «reporter» . El más usado es el luminol. Se analiza la imagen por densitometría. Actualmente, hay programas que permiten la obtención del peso molecular, ¡flipa colega!

Análisis radiactivo (ya no se usa)
Se coloca una película fotográfica contra el Western blot y la actividad radiactiva del marcador que lleva unido el anticuerpo secundario, provoca la aparición en ella de regiones oscuras, que corresponden con las bandas de las proteínas de interés. Alto precio y peligroso para la salud…¡CACA!
Análisis fluorescente
Muy usado. Marcadores fluorescentes que van unidos al anticuerpo secundario, a los cuales se les excita luminicamente, y se detecta su cambio mediante un fotosensor, como una cámara CCD equipado con los filtros de emisión apropiados. Se consigue una imagen digital del Western Blot que puede ser analizada para obtener el peso molecular o la cuantificación proteica. ¡Bueno, bonito, y no tan barato!
Paso 9. Resultado
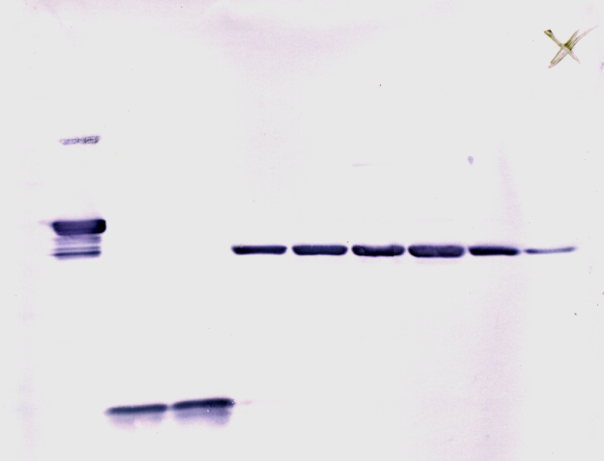
Resultado del Western blot: una membrana con las proteínas separadas en la electroforesis unidas, de las que sólo se disciernen aquellas contra las se ha añadido un anticuerpo. Después de esto podemos escanearlas y estudiarlas de mil maneras diferentes, que no son motivo de esta entrada. (véase la x que marca la orientación)
Por reacción colorimétrica:
Por fluorescencia:
Nota 1: La utilización de hielo en los procesos de electroforesis y electrotransferencia, aporta mejores resultados.
_________________________________________________________________________________
Referencias usadas:
1. http://prezi.com/slsrtiidxrxy/western-blot/
2.https://webfiles.uci.edu/pkaiser/www/KaiserWebpage2010/methods/Yeast%20Protein%20Extracts.htm
3. http://www.slideshare.net/jhojanjralop/extraccion-de-adn-arn-y-proteinas
4. http://www.bioted.es/documentos/ELECTROFORESIS%20BASICA.pdf
5. http://es.wikipedia.org/wiki/Western_blot
7. http://www.decodedscience.com/a-beginner%E2%80%99s-guide-to-gel-electrophoresis/6530
8. http://www.fbmc.fcen.uba.ar/materias/ingen/practicos/proteinas/clases/SDS-PAGE%20Western%20Blot.pdf
________________________________________________________________________________________________________________________________
Bueno, pues después de esta paliza de entrada, que me ha llevado 5 días porque a cada cosa que añadía me perdía más, creo que he conseguido dejar algo bastante claro e útil, por lo menos, yo que tenía que entender el Western Blot, gracias a la creación de esta entrada, lo he entendido, y espero que os sea útil, aunque sea algún detalle, a todos vosotros.
¡Prometo ir editando y mejorando la entrada!
___________________________________________________________________________________________________________________________________
Este post participa en el XXVI Carnaval de Química, que organiza @luisccqq en el blog El Cuaderno de Calpurnia Tate.
¡Ah! Y otra novedad, como viene «que ni pintao» para el tema «Curso express de biologia para verano»…
«Esta entrada participa en el XXIV Carnaval de Biología acogido en Pero eso es otra historia… , que aloja el gran @Ununcuadio en su blog http://worlderlenmeyer.blogspot.com.es/«
¿Nuevos organismos sin clasificar?: Lago Vostok
Hace unos meses se recolectaron unas muestras de perforaciones glaciares en la Antártida, en el Lago Vostok, en concreto perforaciones hasta 3.5 km de profundidad, donde se encuentra este lago.
¿Qué hay de cierto?
En estas muestras de agua, aparte de poder habitar muy pocos organismos, como los microorganismos psicrófilos (para entendernos, bacterias adaptadas a vivir bajo temperaturas extremadamente bajas), se ha encontrado ADN que no pertenece a ninguno de los filo del dominio Bacteria de los que se sabe existencia.
Esto indica que se sabe que es una bacteria pero no pertenece a ningún filo conocido. La cuestión está en que analizando este ADN para intentar hacer un árbol filogenético (Como el de mi compañero J.Fernández, pero intentando relacionar evolutivamente este ADN con el de otras filos conocidos del dominio Bacteria), se ha visto que este ADN no coincide con el de otros microorganismos.
Entonces estamos potencialmente frente a un nuevo filo ,un microorganismo extremófilo por lo que parece ser adaptado a un clima extremo y metabólicamente a altas concentraciones de oxígeno, sobreviviendo también gracias al calor geotérmico.
La controversia de este tema es que no se encontraron organismos vivos al principio, pero poco a poco van teniendo más pruebas que van evidenciando la presencia de estos.
El amarillismo de esta noticia está en que como se supone que como «no están clasificados» juegan con teorías sobre organismos extraterrestres,etc. Pero tranquilos, que por ahora estamos todos a salvo…
Desde Scykness os mantendremos informados sobre las novedades del estudio sobre el Lago Vostok.
Bacteriófagos: Manipulando sistemas inmunes
Hoy en Scykness os queremos contar una de esas curiosidades descubiertas recientemente que bien pueden parecer casi ciencia ficción y que reabren eternos debates…
Primero vamos a definir algo básico de lo que os vamos a contar: Los virus parasitan a las células eucariotas y utilizan su maquinaria de transcripción para replicarse. También tenemos la equivalencia a los virus pero en procariotas, los bacteriófagos o “fagos” para los amigos.

Bien, pues existe un fago que infecta a la bacteria que produce el cólera (Vibrio cholerae), el cual se ha descubierto que es capaz de adueñarse del sistema inmune de la bacteria inactivando este sistema contra los fagos, favoreciendo así la mayor infección y multiplicación de estos.
Un sistema tan sofisticado, específico y efectivo puede ser un verdadero terror para las bacterias. Por una parte solo se ha descubierto que pueda manipular un tipo de sistema inmune que no tienen todas las bacterias llamado “sistema inmune adaptativo”, un sistema basado en la defensa contra fagos con bastante especificidad.
Actualmente existen bacterias que son resistentes a múltiples antibióticos, y la creación de nuevos antibióticos casi no da a basto, una de las terapias que existen como alternativa a los antibióticos son los bacteriófagos, son una respuesta muy específica y efectiva. Esta noticia es muy importante para ello, porque cuanto mejor conozcamos a los fagos más seguras, específicas, y efectivas pueden ser las terapias.
La controversia de todo este tema radica en la respuesta adaptativa de los fagos, que van desarrollando esta técnica de manipulación del sistema inmune de la bacteria con mayor facilidad y con mejor resultado.
Sin duda esto abre otra vez el eterno debate sobre si los virus o fagos son seres vivos o no… ¿Un ente que no esta vivo podría responder adaptativamente y mantener un sistema de manipulación sobre algo? ¿Qué pensáis?